检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
肿瘤微环境是由肿瘤组织中的各种非上皮细胞以及胞外基质共同组成的,其中非上皮细胞主要包括肿瘤浸润的各种免疫细胞、成纤维细胞以及血管内皮细胞等,而肿瘤浸润的免疫细胞又包括T细胞、B细胞、NK细胞和巨噬细胞等。这些细胞通过与癌上皮细胞之间复杂的相互作用影响了肿瘤的发生和进展。近年来单细胞转录组测序等技术已经被广泛应用到肿瘤微环境研究中,但是这些微环境细胞究竟被癌细胞驯化到何种程度,特别是肿瘤微环境细胞是否发生了遗传改变等问题仍然没有得到深入研究。
近年来,多项研究证实了人类正常组织的上皮细胞中普遍存在基因突变,并且“正常”组织上皮细胞中突变的大量积累被认为是一种潜在的癌前病变机制[1]。然而对于大片段的基因组拷贝数变异(somatic copy number alterations, SCNAs)的研究仍然主要依赖大量细胞混合样品的基因组测序(bulk sequencing),这种方式只能检测到频率较高(>5%)的克隆性拷贝数变异,容易忽视潜在的低频率、非克隆性的基因组拷贝数变异,有可能极大地低估了基因组拷贝数变异在人类“正常”组织中的普遍性[2, 3]。

2020年10月22日,js3845金沙线路、北京大学生物医学前沿创新中心(BIOPIC)汤富酬课题组与北京大学第三医院普通外科付卫课题组合作在国际知名学术期刊Cancer Cell上发表题为“Single-cell Multiomics Sequencing Reveals Prevalent Genomic Alterations in Tumor Stromal Cells of Human Colorectal Cancer”的研究论文。该研究从21例微卫星稳定的结直肠癌患者手术切除标本中获取了癌组织、淋巴结转移组织、癌旁组织以及外周血样品,同时从6例非癌症个体中获取了外周血样品,对多种非上皮细胞进行了高精度的单细胞基因组和转录组双组学测序。该研究首先建立并优化了一项单细胞基因组和转录组双组学测序技术,通过对单个细胞的细胞核和细胞质进行分离,使得对同一个单细胞能够同时进行转录组和基因组测序。并在此基础上系统地研究了肿瘤微环境以及正常组织中免疫细胞、成纤维细胞和血管内皮细胞中基因组拷贝数变异的发生频率和特征,揭示了肿瘤微环境中发生基因组拷贝数变异的成纤维细胞存在特异性克隆扩张现象;同时,通过单细胞转录组数据分析发现了5个肿瘤微环境中成纤维细胞特异性表达的肿瘤标志物,均与结直肠癌预后密切相关(图1,2)。
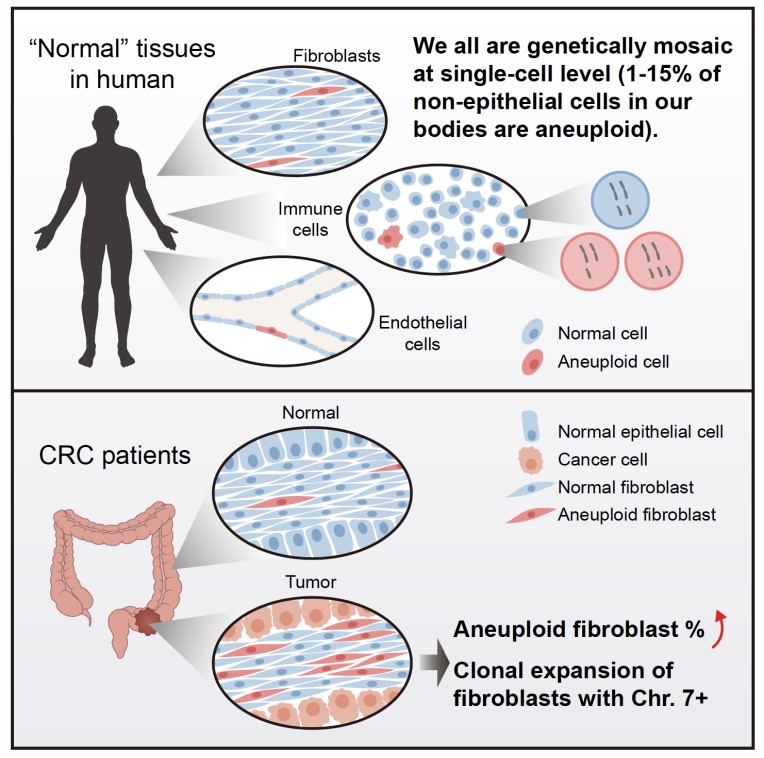
图1. 研究的主要发现概述
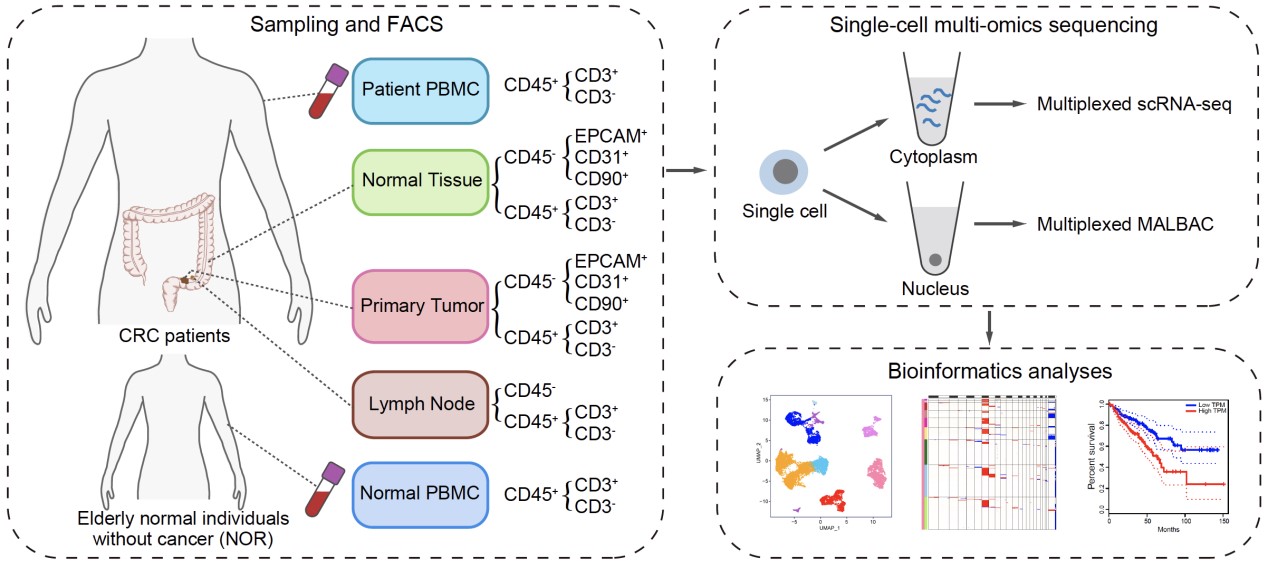
图2. 取样、测序和分析的方案设计
该研究有3个主要发现:
(1)以基因组拷贝数变异为代表的遗传变异在人体正常组织的各种免疫细胞、成纤维细胞以及血管内皮细胞中广泛存在。
在该研究中,研究人员利用单细胞双组学测序技术对来自21例结直肠癌患者以及6例未患癌症的正常人的总计15,312个单细胞的转录组数据进行了深入分析。根据细胞类型特异性表达的标记基因,这些细胞被分为8个主要细胞类群,包括上皮细胞、成纤维细胞、血管内皮细胞、T细胞、NK细胞、B细胞、肥大细胞以及其他髓系免疫细胞。令人惊讶的是,即使在非癌的正常组织中,所有细胞类群中都发现有1%-15%的细胞存在>10 Mb的基因组拷贝数变异。这说明很可能所有人类个体体内的主要细胞类型都存在百分之几的细胞已经发生基因组拷贝数变异等遗传异常,遗传异常不是局限于人体内上皮细胞中的特殊现象,而是所有主要细胞类型中都存在的普遍现象。这些发生了永久的、不可逆的遗传异常的人体细胞有可能参与了人类多种疾病的发生和发展。
通过单细胞基因组数据分析,研究人员发现不仅在肿瘤浸润免疫细胞中存在发生基因组拷贝数变异的遗传异常的免疫细胞(1.3%-11.2%),癌旁“正常”组织浸润的免疫细胞(0.9%-9.7%)、结直肠癌患者的外周血(1.1%-11.5%)以及未患癌症的个体的外周血(2.9%-10.2%)中都存在较高比例的遗传异常的免疫细胞。研究人员一共从8,982个通过严格基因组数据质控的免疫细胞中鉴定出538个(6.0%)发生基因组拷贝数变异的细胞。拷贝数变异在每一位结直肠癌患者(3.0%-11.2%)和每一位未患癌症的正常人(2.9%-10.2%)的免疫细胞中都普遍存在。通过研究发生拷贝数变异的染色体的分布,研究人员发现在女性个体中,整条X染色体的丢失发生频率最高(单个细胞中两条X染色体缺失一条,从XX变为XO),在男性个体中整条Y染色体的丢失发生频率最高(从XY变为XO),而常染色体的拷贝数变异似乎是随机分布的(图3)。有基因组拷贝数变异的血管内皮细胞在癌旁组织(0.0%-1.4%)、结肠肠系膜血管内皮(3.7%-14.9%)和癌组织(3.9%-12.8%)中也都普遍存在。
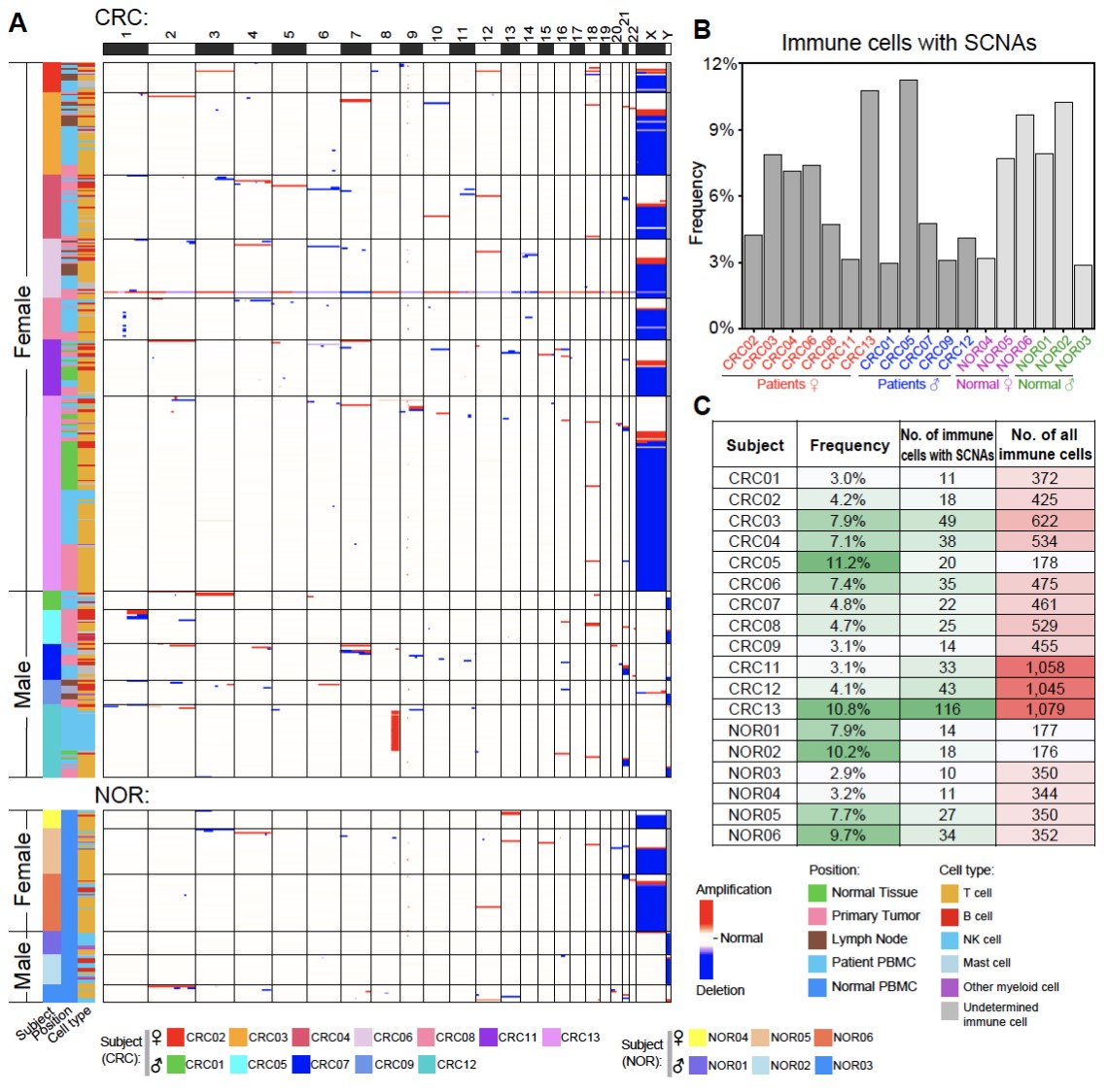
图3. 有基因组拷贝数变异的免疫细胞在不同个体中普遍存在
(2)肿瘤组织中发生遗传异常的成纤维细胞的比例远远高于癌旁正常组织,并且遗传异常的成纤维细胞在肿瘤组织中发生了显著的克隆扩张。
研究人员从来自7例结直肠癌患者癌旁组织的664个成纤维细胞中仅发现37个细胞发生了基因组拷贝数变异,不同患者癌旁组织里成纤维细胞中遗传异常细胞的比例为1.1%-10.6%,而在来自9例结直肠癌患者癌组织的1,183个成纤维细胞中发现了333个发生基因组拷贝数变异的细胞,不同患者肿瘤组织成纤维细胞中遗传异常细胞的比例为11.1%-47.7%,相比于癌旁 “正常”组织比例增加了4倍左右(图4)。而且癌旁组织中成纤维细胞的基因组拷贝数变异呈现相对随机的分布特征,而肿瘤组织成纤维细胞中显著富集7号染色体拷贝数增加的细胞。在88个来自肿瘤组织发生7号染色体拷贝数变异的成纤维细胞中,有85个细胞存在整条7号染色体拷贝数增加,并且这85个细胞中位于7号染色体上的基因的平均表达水平要显著高于(增加了26%)没有基因组拷贝数变异的成纤维细胞。7号染色体拷贝数增加的特异性富集提示了肿瘤微环境中发生基因组拷贝数变异的遗传异常的成纤维细胞的克隆扩张(图5)。这说明肿瘤微环境细胞不是简单地接受癌上皮细胞的“诱导”而被动地参与肿瘤发生过程,而是已经发生不可逆的、永久的遗传变异,因而很可能主动地促进了癌上皮的肿瘤发生过程。遗传变异不是像原来普遍认为的那样只发生在肿瘤上皮细胞中,而是也广泛发生在非上皮的肿瘤微环境细胞中。
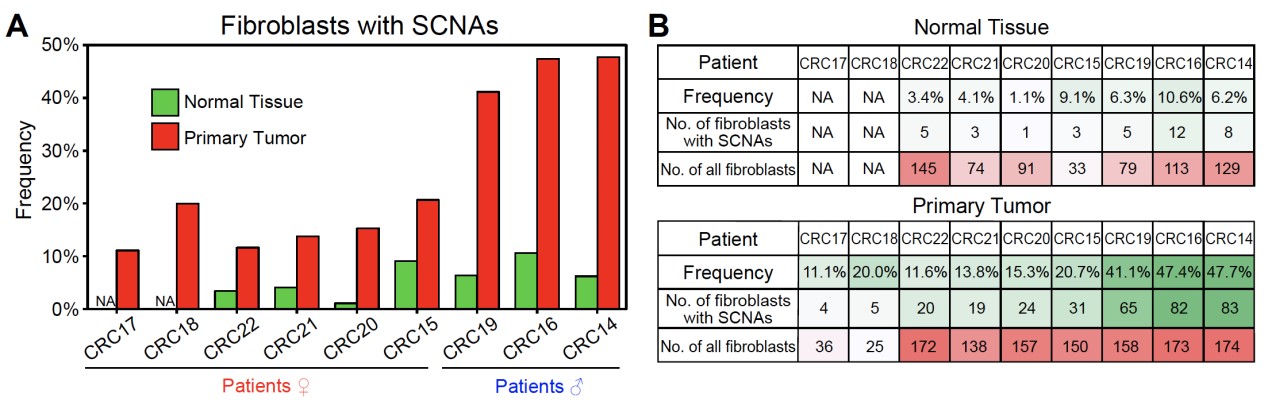
图4. 发生基因组拷贝数变异的成纤维细胞在肿瘤组织中的比例远远高于在癌旁“正常”组织中的比例
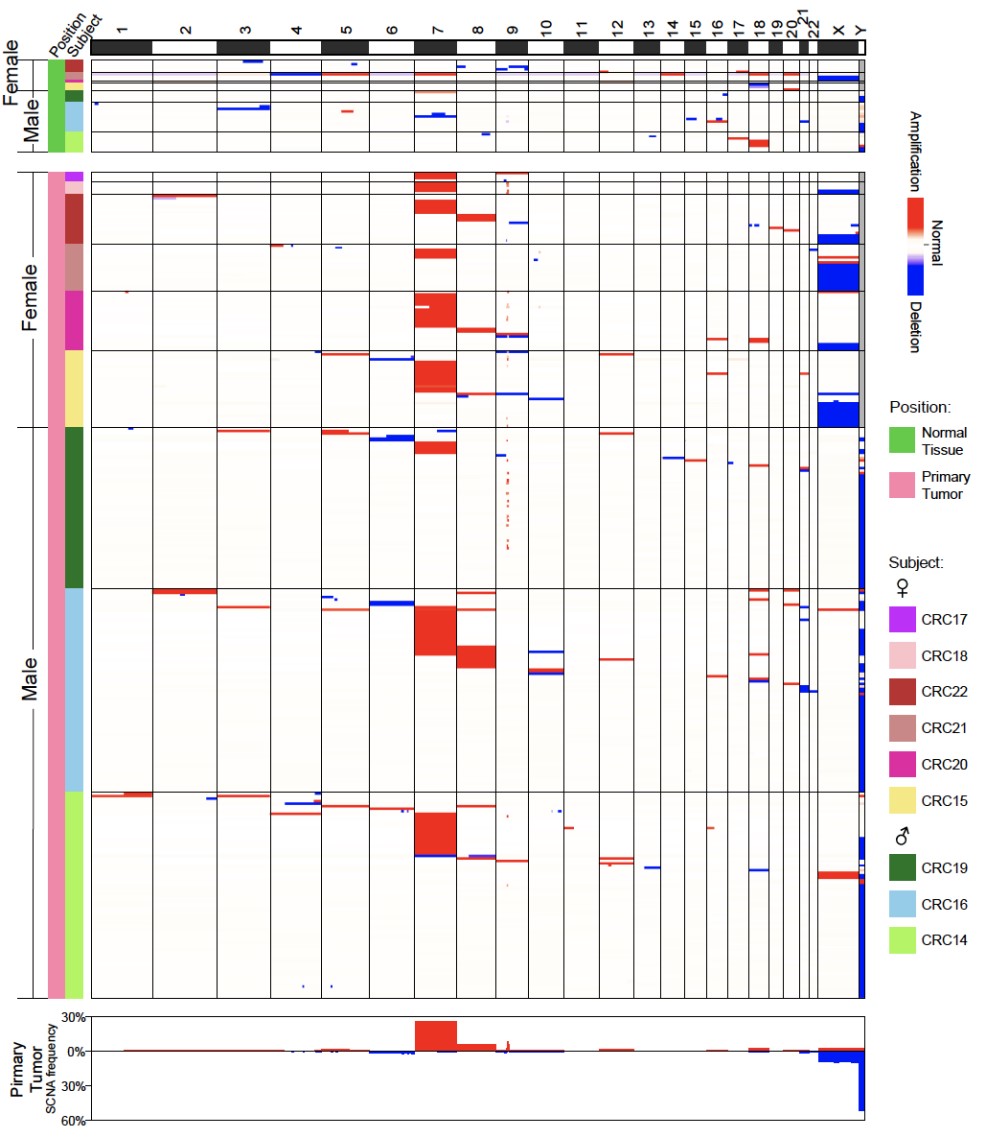
图5. 肿瘤组织中的成纤维细胞显著富集7号染色体拷贝数增加的细胞
(3)发现了5个肿瘤微环境中成纤维细胞特异表达的肿瘤标志基因,它们的表达水平均与结直肠癌预后密切相关。
研究人员鉴定出肿瘤组织中成纤维细胞相对于癌旁组织中成纤维细胞差异表达的76个基因,包括59个上调基因和17个下调基因。其中BGN、RCN3、TAGLN、MYL9和TPM2在肿瘤组织成纤维细胞中明显上调,经TCGA数据库验证,其高表达与结直肠癌预后差明确相关。免疫组化实验进一步验证了这5个基因在结直肠癌肿瘤微环境成纤维细胞中特异性高表达,确定了这5个基因是肿瘤微环境中成纤维细胞特异性表达的肿瘤标志基因(图6)。这一发现拓展了肿瘤标志基因的种类,从原来主要聚焦于癌上皮细胞表达的标志基因变成同时关注肿瘤微环境细胞中特异性表达的肿瘤标志基因。而且肿瘤微环境细胞特异性表达的肿瘤标志基因更有可能具有跨不同癌种的普适性,其临床应用场景可能更广泛。
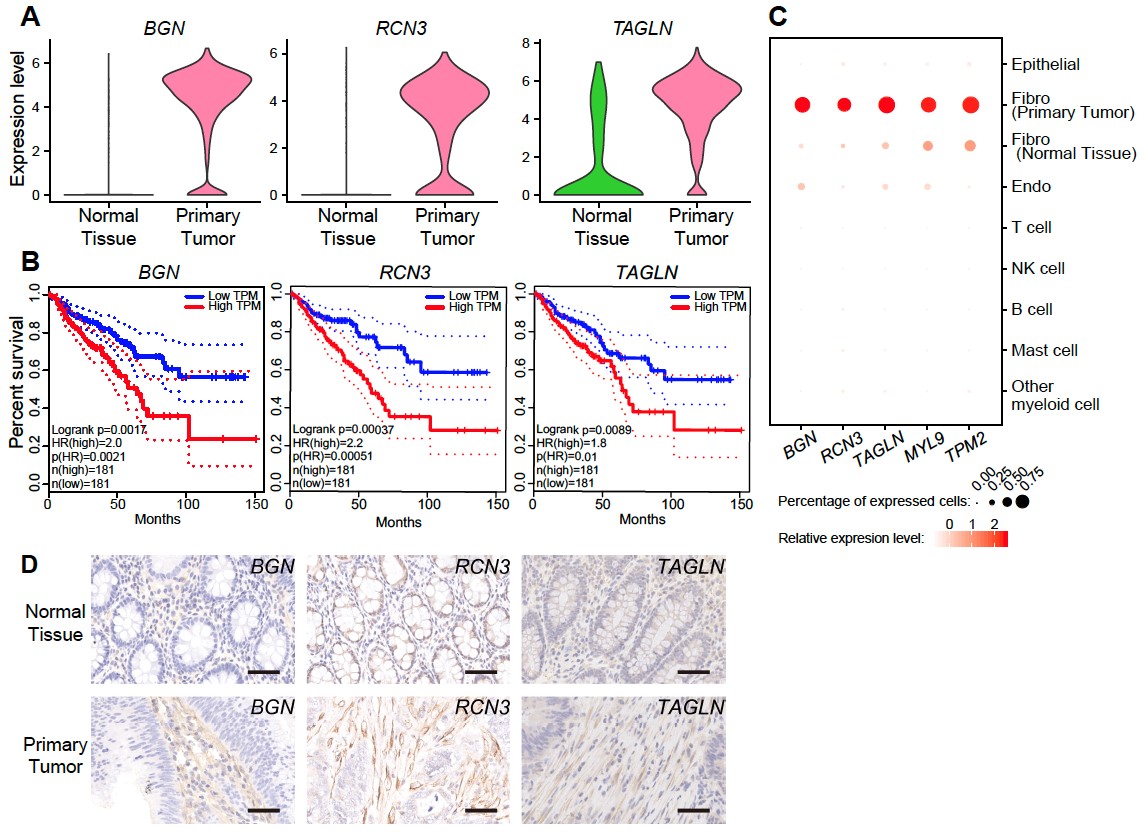
图6. 与结直肠癌预后密切关联的成纤维细胞特异性表达的肿瘤标志基因
综上所述,本项研究利用高精度单细胞多组学测序技术,深入系统地分析了27个人类个体的多种非上皮细胞的基因组拷贝数变异情况,揭示了基因组拷贝数变异普遍存在于人体成纤维细胞、免疫细胞和血管内皮细胞中,并发现了结直肠癌肿瘤微环境中的成纤维细胞中发生基因组拷贝数异常细胞的比例大幅度增加,而且这类遗传异常的成纤维细胞在肿瘤组织中存在克隆扩张现象。此项研究突破了人们对人体“正常”组织中细胞遗传变异的认知,充分证明了以基因组拷贝数变异为代表的遗传变异在人群中普遍存在,在人体主要细胞类型中普遍存在,提示基因组拷贝数变异在人类疾病和衰老中可能存在重要作用,并对肿瘤微环境和肿瘤治疗研究提供了新的线索。
北京大学生物医学前沿创新中心(BIOPIC)博士生周圆、卞舒惠博士(现为生殖医学国家重点实验室PI)、博士生崔月利以及北京大学第三医院普通外科周鑫主治医师为该论文的并列第一作者。北京大学未来基因诊断高精尖创新中心汤富酬教授和北京大学第三医院普通外科付卫教授为该论文的共同通讯作者。该研究项目得到了国家自然科学基金和北京未来基因诊断高精尖创新中心(ICG)的支持。
原文链接:https://www.cell.com/cancer-cell/fulltext/S1535-6108(20)30489-X
参考文献:
1. Yizhak, K. et al. RNA sequence analysis reveals macroscopic somatic clonal expansion across normal tissues. Science 364, aaw0726, (2019).
2. Jacobs, K. B. et al. Detectable clonal mosaicism and its relationship to aging and cancer. Nat Genet 44, 651-658, (2012).
3. Laurie, C. C. et al. Detectable clonal mosaicism from birth to old age and its relationship to cancer. Nat Genet 44, 642-650, (2012).